




Polytänchromosomen mit einem Querbandenmuster wurden bei Pflanzen erstmalig von Nagl in den großen Zellen des Suspensors von Phaseolus coccineus und P. vulgaris entdeckt (Nagl 1962a; Nagl 1969b). Zwar kommen Chromosomen mit der Tendenz zur Polytänie auch in sechs weiteren von insgesamt zehn untersuchten Phaseolus-Arten vor, aber keine wies bisher einen höheren Polyplodisierungsgrad und eine solche kompakte Bündelung auf wie die von P. coccineus (vgl. Nagl 1974, Tabelle 3). Kleinere und weniger stark gebündelte Polytänchromosomen wurden auch im Tapetum der Antheren von P. coccineus und Vigna unguiculata beobachtet (Guerra und Carvalheira 1994).
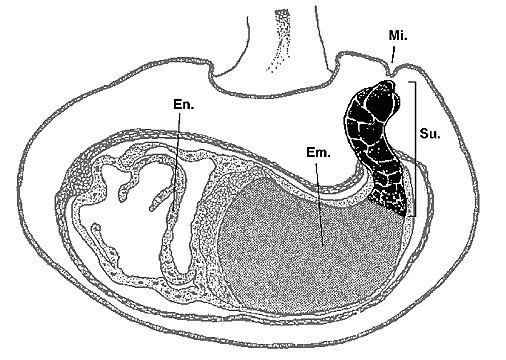
Der Suspensor oder Embryoträger ist bei Angiospermen ein kurzlebiges, stielartiges Gebilde, das aus einem Teil des Proembryo hervorgeht. Die Aufgabe des Suspensors ist es, den Embryo während seiner Entwicklung in das sich bildende Nährgewebe hineinzuschieben und ihn mit Nährstoffen und Wachstumsfaktoren zu versorgen (Übersicht bei Yeung und Meinke 1993). Danach degeneriert er und läßt sich in reifen Samen nicht mehr nachweisen.
In Samen von Phaseolus besteht der Suspensor aus einem Verband von einigen hundert Zellen deren Größe vom Embryo zur Mikropyle hin zunimmt. Die größten Zellen des Suspensors befinden sich im basalen Teil, in direkter Nachbarschaft zur Mikropyle (Abbildung 1). Struktur und Ultrastruktur des Suspensors während der Embryogenese wurden sowohl lichtmikroskopisch als auch elektronenmikroskopisch ausführlich untersucht (Schnepf und Nagl 1970; Yeung und Clutter 1978; 1979).

Da das Volumen der Suspensorzellen von P. coccineus direkt mit ihrem DNA-Gehalt korreliert, wurde aus dem maximal beobachteten Volumen der größten Zellen auf einen maximalen relativen DNA-Gehalt von 4096 n geschlossen (Nagl 1962a). Spätere Messungen an Feulgen-gefärbten Präparaten von Polytänchromosomen ergaben einen noch höheren Maximalwert von 8192 C (Brady 1973). Es zeigte sich auch, daß nicht alle großen und basal gelegenen Zellen des Suspensor den selben hohen DNA-Gehalt aufweisen, sondern ihre Werte zwischen 256 C und 8192 C schwanken.
Der hohe DNA-Gehalt kommt dadurch zustande, daß die Kerne der Suspensorzellen etwa 10 bis 12 Endoreduplikations-Zyklen durchlaufen. Die Endoreduplikation (vgl. Pearson 1974) stellt eine Abkürzung des normalen mitotischen Zellzyklus dar, bei dem die DNA ohne strukturelle Veränderungen des Zellkerns dupliziert wird, wie sie für eine Mitose typisch wären (Levan und Hauschka 1953). Im Fall von Polytänchromosomen werden dadurch zahlreiche Chromatiden (in diesem Zusammenhang nach Nagl (Nagl 1978) Endochromosomen genannt) gebildet, die sich nicht voneinander trennen, sondern Seite an Seite beieinander liegen bleiben. Daß die Endochromosomen bei pflanzlichen Polytänchromosomen allerdings nicht so genau parallel ausgerichtet sind wie bei den klassischen Polytänchromosomen, läßt sich, nach Nagl, auf die Zerstäubungs- oder Z-Phase (Heitz 1929) zurückführen. Sie hat zur Folge, daß die Polytänchromosomen von Pflanzen normalerweise kaum oder keine Querbandenmuster ausbilden (Zur Ausbildung unter besonderen Zuchtbedingung, siehe unten).
Für eine Gesamtübersicht der Arbeiten an den Polytänchromosomen von Phaseolus sei auf zwei ausführliche Zusammenfassungen verwiesen (Nagl 1974; Nagl 1981). An dieser Stelle sollen nur die Aspekte der strukturellen Charakterisierung herausgegriffen werden, die zur bisherigen Identifizierung der Polytänchromosomen beigetragen haben.
Bereits die erste Beschreibung der Polytänchromosomen von Phaseolus coccineus zeigte, daß die elf Chromosomenpaare einen ähnlichen Grundaufbau haben (Nagl 1962b). Alle elf besitzen in der Regel im medianen Abschnitt einen Bereich aus Heterochromatin, dem die beiden mehr oder weniger verschieden langen Arme aus Euchromatin anliegen. Innerhalb des heterochromatischen Bereiches wird die Spindelansatzstelle (Centromer) angenommen, die manchmal als Lücke sichtbar ist. Im Euchromatin der Arme wurden in unterschiedlicher Entfernung vom Centromer zusätzlich lockere heterochromatische Zonen beobachtet, die als "Querscheiben" bezeichnet wurden. Anhand des Verteilungsmusters von eu- und heterochromatischen Zonen sowie den "Querscheiben" ließen sich zunächst vier der elf Chromosomenpaare (Typ A bis D) individuell ansprechen (Nagl 1962b).
Kurze Zeit später wurden zwei Chromosomen S1 und S2 identifiziert, die mit dem Nukleolus bzw. den Nukleolen in Verbindung stehen (Nagl 1965). Nach Nagl wiesen diese beiden SAT-Chromosomen je einen "Endosatelliten" auf, der über fadenartige Chromatinstränge, die sich durch die Nukleolus-organisierende Region (NOR) ziehen, mit dem Chromosom verbunden ist. Ferner fiel bei diesen Untersuchungen auf, daß die Querscheiben weniger deutlich ausgeprägt waren als bei vorangegangenen Beobachtungen. Dies wurde als Veränderung des Chromosomenbaus in Präparaten aus verschiedenen Jahren gedeutet (vgl. Hasitschka-Jenschke 1961).
Weitere Untersuchungen zeigten, daß sich alle elf Chromosomenpaare anhand ihrer verschiedenen Längen, Armlängenverhältnisse und Verteilung der heterochromatischen Abschnitte mehr oder weniger sicher identifizieren ließen (Nagl 1967). Nagl (1967) stellte ein vorläufiges Idiogramm aller Polytänchromosomen von Phaseolus coccineus vor und benannte die Chromosomen mit den römischen Ziffern I bis XI. Gleichzeitig wurde aber auch darauf hingewiesen, daß sowohl die Längen als auch das Heterochromatinmuster einer gewissen Variabilität unterliegen. Diese behindert die regelmäßige Identifizierung und führt dazu, daß sich nicht alle Chromosomen mit der gleichen Sicherheit ansprechen lassen. Insbesondere durch die Streckung der Chromosomen kann ein ursprünglich kürzerer Arm zum längeren werden. Der Vergleich mit mitotischen Metaphasechromosomen zeigte ferner, daß das kleinste und das größte Polytänchromosom (I, XI) jeweils etwa 30 mal länger sind ist das entsprechende Metaphasechromosom (s. Abschnitt 1.5). Dieser Wert wurde auch mit dem Verlängerungsfaktor von Pachytänchromosomen gegenüber mitotischen Metaphasechromosomen verglichen. Aus den Messungen von Krishan und De (1965) bei Phaseolus aureus wurde für Pachytänchromosomen ein Verlängerungsfaktor von 21 berechnet, der deutlich unter dem Faktor 30 der Polytänchromosomen liegt (Nagl 1967).
An Polytänchromosomen der nahe verwandten Art P. vulgaris wurde festgestellt, daß sich durch zweiwöchiges Absenken der Anzuchttemperatur eine reversible Bildung von Querscheiben/-banden induzieren ließ. Doch obwohl sich, je nach Länge der Chromosomen, zwischen 40 bis 160 Querbanden beobachten ließen (Nagl 1969a), wurde dieses Muster von Querscheiben nicht zur weiteren Identifizierung herangezogen.
Zu Beginn der 80er Jahre wurden Markierungsmethoden, Bänderungsmethoden und die Technik der in situ-Hybridisierung (s. Abschnitt 1.4.2.3) entwickelt, von denen die meisten bis heute eine wichtige Rolle bei der Charakterisierung von Chromosomen spielen.
Die ersten und umfangreichen Markierungen an Polytänchromosomen von Phaseolus mit verschiedenen radioaktiv markierten Nukleinsäuren und Aminosäuren sowie unterschiedliche cytochemische Färbungen wurden von Avanzi et al. (1970) durchgeführt. Sie leistete aber zur strukturellen Charakterisierung im Sinne einer individuellen Identifizierung der Chromosomen keinen maßgeblichen Beitrag.
Größere Relevanz hatten die Versuche, Guanin-reiche Abschnitte auf den beiden SAT-Chromosomen von P. coccineus mittels radioaktiv-markiertem Actinomycin D (AMD) nachzuweisen (Cionini und Avanzi 1972). Die Markierung an den beiden SAT-Chromosomen I und V zeigte sich am stärksten im Nukleolusbereich sowie auf den heterochromatischen Bereichen am Centromer. Überraschenderweise blieben jedoch einzelne Abschnitte des Heterochromatins unmarkiert und umgekehrt zeigten manche euchromatische Bereiche bereits nach kurzer Zeit eine deutliche Markierung. Später folgten weitere Versuche zur Untersuchung der DNA-Methylierung, die deutlich machten, daß das centromerische Heterochromatin der am stärkste methylierte Bereich der Polytänchromosomen ist (Frediani et al. 1986; Andreucci et al. 1994). Brady und Clutter (1974) wiesen bei ihren Untersuchungen zur Strukturveränderung der Polytänchromosomen während des Replikationszyklus darauf hin, daß die sog. [beta]-heterochromatischen Bereiche (die heterochromatischen Bereiche um das Centromer) im aufgelockerten Zustand der Replikation nicht vom Euchromatin zu unterscheiden seien. Das Heterochromatin des Centromers wurde im übrigen von den Autoren, in Anlehnung an die Polytänchromosomen der Dipteren, als [alpha]-Heterochromatin angesprochen. Diese Art der Bezeichnung hat sich aber nicht durchgesetzt.
Chromosomen-Bänderungs-Methoden beruhen im Prinzip darauf, daß sich einige Abschnitte von Chromosomen (evtl. nach geeigneter Vorbehandlung) stärker anfärben lassen als andere. Daraus ergibt sich ein Muster, anhand dessen sich die Chromosomen unter Umständen individuell erkennen lassen. Allgemeine Verwendung in der Cytogenetik finden vor allem das "C-Banding" (Arrighi und Hsu 1971) und "G-Banding" (Schnedl 1971; Sumner et al. 1971) sowie Fluorochrome mit gewisser Bindungspräferenz zu AT- oder GC-reichen Sequenzen: Hoechst 33258 (AT) (Hilwig und Gropp 1972) und DAPI (AT) (Dann et al. 1971), Quinacrin-dihydrochlorid bzw. -Mustard (Caspersson et al. 1969) und Chromomycin A3 (GC) (Schweizer 1976c; Schweizer 1976a).
Um auf den Polytänchromosomen von P. coccineus und P. vulgaris ein differentielles Bänderungsmuster zu erzeugen, nutzte Schweizer (1976b) die C-Bänderung und die Fluorochrome Chromomycin A3 (CMA) bzw. DAPI in Kombination mit Actinomycin D (AMD). Die gleichen Färbungen führte er auch an Metaphasechromosomen durch und verglich die Bänderungsmuster mit denen der Polytänchromosomen. Die C-Bandenmuster der beiden Chromosomen-Typen stimmten im wesentlichen überein, wiesen aber bei den Polytänchromosomen aufgrund ihrer Größe eine bessere Auflösung auf. Insbesondere zeigten sich im Centromerbereich der Polytänchromosomen mehrere Blöcke oder Scheiben, wohingegen bei den Metaphasechromosomen am Centromer nur eine einzige Bande beobachtet werden konnte. Die Färbung mit CMA und DAPI/AMD zeigte zwei unterschiedliche Typen von Heterochromatin bei den Polytänchromosomen. Der eine Typus färbte sich stark mit CMA aber nur schwach mit DAPI/AMD an, während sich der andere umgekehrt dazu verhielt. Außerdem fiel auf, daß sich die SAT-Chromosomen aufgrund ihrer Variablität nicht eindeutig identifizieren ließen und auch nicht mit ihren entsprechenden Metaphasechromosomen in Einklang zu bringen waren.
Aufgrund der zwei Paare von SAT-Chromosomen bei P. coccineus (Nagl 1965) ging man von vier Nukleolus-organisierenden (NO) Chromosomen aus, obwohl es Hinweise gab, daß ihre tatsächliche Anzahl höher liegt (Nagl 1965; Schweizer 1976b). Untersuchungen an Polytänchromosomen und Metaphasechromosomen mit Silberfärbung und Giemsa-C-Bänderung zeigten schließlich, daß P. coccineus tatsächlich sechs NO-Chromosomen besitzt (Schweizer und Ambros 1979), was später nochmals durch die Ergebnisse der in situ-Hybridisierungen bestätigt wurde (s. Abschnitt 1.4.2.3).
Um die Struktur der beiden NO-Chromosomenpaare I und V während der Samenentwicklung genauer zu studieren (Forino et al. 1979), wurden die Chromosomen-Abschnitte erstmalig nach einer festgelegten Nomenklatur beschrieben (Paris Conference 1972). Die Polytänchromosomen wurden danach in Regionen unterteilt, die Banden numeriert und der kurze bzw. lange Arm der Chromosomen mit den Symbolen "p" bzw. "q" gekennzeichnet. Weitere Analysen gaben Hinweise auf eine unterschiedliche Ausprägung, selbst zwischen Homologen (Cionini et al. 1982) und verschiedenen Polytänchromosomen (Tagliasacchi et al. 1983).
Die Technik der in situ-Hybridisierung (ISH) wurde vor 29 Jahren etwa zeitgleich von drei Arbeitsgruppen entwickelt (Gall und Pardue 1969; John et al. 1969; Buongiorno-Nardelli und Amaldi 1970). Die ISH machte es möglich, markierte DNA- oder RNA-Sequenzen als Sonden zu verwenden und ihr Vorkommen im Genom direkt "an Ort und Stelle" (in situ) auf dem Chromosom nachzuweisen. Die Auswahl an Sonden-DNA/RNA war anfangs auf Satelliten-DNA und ribosomale RNA beschränkt, die durch physikalische Trennung gewonnen werden konnten. Erst die Vervielfältigung nahezu beliebiger DNA-Fragmente durch molekularbiologische Methoden wie z. B. die Klonierung in Plasmid-Vektoren (Bolivar et al. 1977) oder die Polymerase Kettenreaktion (PCR) (Saiki et al. 1985; Mullis et al. 1986) erlaubten es, auch andere DNA/RNA-Sequenzen in ausreichender Menge zu produzieren und daraus markierte Sonden herzustellen.
Lange Zeit war die Markierung der Sonden mit radioaktiven Isotopen wie z. B. 3H oder 125I und deren Detektion durch Autoradiographie die einzige Nachweismethode. Sie ist heute von den verschiedenen Varianten der nichtradioaktiven ISH abgelöst. Bei ihr wird in die Sonden-DNA/RNA entweder einzelne Nukleotide mit Reportermolekülen (z. B. Biotin, Digoxigenin) eingebaut ("Markierung") oder die Sonde wird als ganzes chemisch modifiziert. Die Detektion der Reportermoleküle bzw. der modifizierten Sonde erfolgt mit Antikörpern oder dem Protein Avidin (bzw. Streptavidin oder Derivaten von beiden), die jeweils mit einem Fluoreszenzfarbstoff oder einem Enzym gekoppelt sind (indirekte Detektion). Während die Fluorochrome eine umgehende Untersuchung im Fluoreszenzmikroskop ermöglichen, ist für den Nachweis mittels enzym-gekoppelter Verfahren eine zusätzliche Reaktion notwendig, um ein farbiges Präzipitat zu erhalten. Eine noch schnellere Detektion erlaubt die Verwendung von Sonden, in die bereits bei der Markierung Fluoreszenzfarbstoffe eingebaut werden (direkte Detektion). Basierend auf diesen Grundtechniken wurden zahlreiche Varianten mit unterschiedlichen Reportermolekülen, Fluorochromen, Antikörpern und Enzymen entwickelt (Übersichten bei Lichter et al. 1991; Speel et al. 1995). Die am häufigsten verwendete Variante ist die Fluoreszenz-in situ-Hybridisierung (FISH) mit indirekter oder direkter Detektion (vgl. McNeil et al. 1991; Trask 1991). Mit der FISH wurden bis dato Fragestellungen auf den verschiedensten Gebieten wie z. B. numerische und strukturelle Chromosomenaberrationen, Kartierung von Genen und DNA-Fragmenten, Aufbau der Kernstruktur, Struktur und Evolution von Chromosomen bis hin zur DNA-Replikation und Gen-Amplifikaton untersucht (Heng et al. 1997). Dabei spielte oftmals auch die Möglichkeit mehrere Sonden gleichzeitig und in unterschiedlichen Fluorochromen (oder Mischungen von Fluorochromen) darzustellen, eine wichtige Rolle (Le Beau 1996).
Die ersten Versuche mittels FISH erlaubten allgemein nur den Nachweis von repetitiven Sequenzen wie z. B. Satelliten-DNA oder die Gene der 18S-25S und 5S RNAs (z. B. Baumann et al. 1981; Langer-Safer et al. 1982; Manuelidis et al. 1982). Die Sensitivität der indirekten Detektion wurde jedoch gesteigert, um auch kürzere und nur in wenigen Kopien vorliegende Zielsequenzen, nachweisen zu können (Viegas-Pequignot et al. 1989). Dazu wird entweder die Zahl der am Nachweis beteiligten Fluorochrom-Moleküle direkt im Präparat vervielfältigt und/oder die schwachen Fluoreszenzsignale mittels hochempfindlicher CCD-Kameras aufgenommen (Ballard und Ward 1993; Ferguson et al. 1996).
Um die Zahl der Fluorochrom-Moleküle im Präparat zu erhöhen (Signalamplifikation) gibt es, je nach Reportermolekül das zum Markieren der Sonde verwendet wurde, im wesentlichen zwei gängige Methoden: Sonden, die mit Biotin markiert und über Fluorochrom-gekoppeltes Avidin detektiert werden, inkubiert man anschließend mit einem Anti-(Strept)Avidin-Antikörper der mit Biotin konjugiert ist. Im nächsten Schritt inkubiert man mit Fluorochrom-gekoppeltem Avidin (Pinkel et al. 1986). Sonden, die mit anderen Reportermolekülen wie z. B. Digoxigenin (DIG) markiert wurden, inkubiert man mehrfach mit einem anderen, jeweils passenden Fluorochrom-gekoppelten Antikörper, so daß eine Kaskade entsteht. Auf weitere Detektionsmöglichkeiten mittels enzymatischer Umsetzung von Fluorochrom- bzw. Reportermolekül-konjugierten Substraten (Raap et al. 1995; Kagiyama et al. 1995; Paragas et al. 1997) oder Chemilumineszenz (Musiani et al. 1998) soll hier nicht eingegangen werden.
Die Anwendung der ISH an Chromosomen von Pflanzen stand hinter der an Chromosomen von Tieren zurück (Übersichten bei: Jiang und Gill 1994; Lavania 1998). Die erste nichtradioaktive ISH wurde erst im Jahr 1985, drei Jahre nach den Untersuchungen an Polytänchromosomen von Drosophila und Metaphasechromosomen von Mäusen veröffentlicht (Rayburn und Gill 1985). Der Hauptgrund ist, daß die Anforderungen, die an die Präparate für nichtradioaktive ISH gestellt werden müßen, für Chromosomenpräparate aus pflanzlichen Zellen nur unter erheblichem Aufwand und mit viel Erfahrung zu erfüllen sind (Hinnisdaels et al. 1994). Die Bedingungen sind viele nicht überlappend liegende Chromosomen, die weder von der Zellwand überdeckt sind, noch auf Resten von Cyto-, oder wie hier bei Polytänchromosomen, Nukleoplasma liegen.
Wie bei der ISH an tierischen Chromosomen, so ist auch die Detektion von low-copy Sequenzen bei pflanzlichen Chromosomen sehr schwierig. Bei allen bisherigen Berichten über den Nachweis von Sequenzen in geringer Kopienzahl oder gar einmaligen Sequenzen betrug die Länge der Zielsequenz immer mehr als 10 kbp (siehe Ausführungen bei Jiang und Gill 1994). Wie bei den Untersuchungen an tierischen Chromosomen hat sich auch bei der Anwendung an pflanzlichen Chromosomen die FISH mit all ihren Varianten (GISH, Multicolor-FISH, Interphase FISH) als eine Standardmethode bei der Genom-Kartierung durchgesetzt (Jiang und Gill 1996).
An den Polytänchromosomen von Phaseolus coccineus wurde die erste ISH mit 3H-markierter ribosomaler 18S-5,8S-25S RNA als Sonde durchgeführt (Avanzi et al. 1972; Brady und Clutter 1972). Brady und Clutter beobachteten Signale nur auf den beiden SAT-Chromosomenpaaren I und V. Avanzi et al. hingegen fanden Signale auf insgesamt drei Paaren: I, V und zusätzlich II. Eine Anzahl von drei Chromosomen mit Loci der 18S-25S rDNA konnte auch von anderen Autoren unter Verwendung von radioaktiver ISH sowie FISH bestätigt werden (Schumann et al. 1990; Tagliasacchi et al. 1993; Nenno et al. 1994). Desweiteren wurde die Verteilung der Gene für die ribosomale 5S RNA untersucht, und ihre Position auf den Chromosomen I und VI bestimmt (Durante et al. 1977).
Durante et al. (1987) untersuchten an Polytänchromosomen die Verteilung von zwei hochrepetitiven DNA-Fraktionen, die durch isopyknische Zentrifugation gewonnen wurden. Eine Fraktion bestand aus fast renaturating sequences (FRS), die 20,8% der gesamten DNA ausmachten und die zweite Fraktion aus Satelliten-DNA mit der Dichte 1,702 g/ml. Um Mißverständnissen vorzubeugen sei darauf hingewiesen, daß der Ausdruck "Satelliten-DNA" nichts mit dem "Satelliten" eines Chromosoms zu tun hat (vgl. Rieger et al. 1991). Die stärksten Signale beider Fraktionen lagen präferentiell in den heterochromatischen Bereichen um das Centromer. Zusätzlich waren sie im interkalaren Heterochromatin sowie an den Enden mancher Chromosomen lokalisiert. Die Signalintensität in den heterochromatischen Regionen war dabei variabel. Auffallend war, daß der mediane Bereich von Chromosom II, der im Idiogramm von 1967 als heterochromatisch beschrieben worden war, weder Signale mit den FRS noch mit der Satelliten-DNA aufwies. Die Autoren machten darauf aufmerksam, daß eben dieser Bereich, wie auch die interkalaren Heterochromatinbereiche auf den anderen Chromosomen, eher dekondensiert erscheint und daher mehr den Eindruck von Eu- als von Heterochromatin macht.
Als erste low-copy Sequenzen bei P. coccineus konnten die Gene des Hauptspeicherproteins Phaseolin mittels radioaktiver ISH auf Chromosom 3 lokalisiert werden (Schumann et al. 1990). Bald darauf folgte der Nachweis von low-copy Genen des Polygalacturonase-hemmenden Proteins (PGIP) bei P. vulgaris auf Chromosom X (Frediani et al. 1993). Nach Frediani entspricht das Idiogramm der Polytänchromosomen von P. vulgaris dem von P. coccineus.
In der ersten FISH an Polytänchromosomen von P. coccineus wurde eine humane Telomer-Sequenz als Sonde eingesetzt, die die Enden der Polytänchromosomen markierte (Nagl 1991). Versuche mit anderen repetitiven Sequenzen, wie z. B. der 18S-25S rDNA, zeigten aber nur vergleichsweise schwache Fluoreszenzsignale. Erst mit der Einführung der Pepsin/HCl-Vorbehandlung für die Präparate der Polytänchromosomen ließ sich die Signalintensität der 18S-25S rDNA-Signale steigern und die Loci auf den drei NO-Chromosomen nachweisen (Nenno et al. 1994). Darüber hinaus ermöglichte die Vorbehandlung mit der Pepsin/HCl erstmals auch die nichtradioaktive Detektion der low-copy Gene von Phaseolin mittels FISH, die zuvor nur mittels radioaktiver ISH nachgewiesen werden konnten.




