




Die Durchführung der Hybridisierung mit den Sonden für die 5S und 18S-25S ribosomalen RNA- sowie Phaseolin-Gene erfolgte gemäß dem Protokoll von Nenno (1992) und Nenno et al. (1994).
Die Ausbildung von DNA-Doppelsträngen aus zwei komplementären Einzelsträngen ist ein reversibler Prozess, der von verschiedenen Faktoren beeinflußt wird. Diese sind der prozentuale Gehalt der Basen Guanin und Cytosin (%GC), die Länge des DNA-Doppelstranges (n), die Konzentration monovalenter Kationen (z. B. [Na+]) und ggf. Doppelstrang-destabilisierender Agenzien (z. B. Formamid). Miteinander stehen diese Faktoren nach einer empirisch ermittelten Formel in Zusammenhang (Meinkoth und Wahl 1984). Mittels dieser Formel läßt sich die Schmelztemperatur (temperature of melting, Tm) eines Doppelstranges berechnen.
In der praktischen Anwendung im Laufe der letzten Jahre hat sich allerdings gezeigt, daß die nach dieser Formel berechnete Schmelztemperatur nicht als absolut anzusehen ist, sondern nur einen geeigneten Anhaltspunkt liefert. Vermutlich verhalten sich DNA-Doppelstränge in situ nicht so wie in Lösung (Leitch et al. 1994).
Nach Britten und Kohne (1968) läßt sich aus der mit (1) errechneten Schmelztemperatur die optimale Hybridisierungstemperatur berechnen.
Setzt man in (1) einen GC-Gehalt von 39,3% für Phaseolin, eine Natrium-Konzentration von 0,39 M, eine Länge von ca. 200 Basenpaare (gängige angenomme Fragmentgröße nach Nick-Translation) und 50% Formamid ein, so erhält man eine Schmelztemperatur (Tm) von 58°C und aus (2) eine Hybridisierungstemperatur (Th) von 33°C. Unter Berücksichtigung dieser Richtwerte wurden die Sonden aus klonierten Sequenzen unter den genannten Bedingungen bei einer Temperatur von 37°C hybridisiert, was als hoch stringent anzusehen ist.
Material, Chemikalien und Lösungen
Fixogum: Montagekleber (Marabu 290117000)
20 x SSC: 3 M NaCl, 0,3 M Na3-Citrat*2H20, pH 7,0
Formamid-Lösung: 70% deionisiertes Formamid (Biomol) in 2 x SSC
kalte Ethanolreihe: 70% und 85% Ethanol, 100% Ethanol abs., lagern bei
-20°C
Dextransulfat: 50% (w/v) in Aqua dest.
Denaturierung und Hybridisierung
Präparate |
Hybridisierungslösung |
|---|---|
- Präparate 4 min in Formamid-Lösung bei 70°C denaturieren |
|
- Präparate je 5 min/Stufe in -20°C kalter Ethanolreihe entwässern. - OT für ca. 1 min in einen OT-Halter stellen und Ethanol ablaufen lassen |
- Pro OT 10 µl Hybridisierungslösung in einem Reaktionsgefäß mischen:
5 µl 100% (v/v) deionisiertes Formamid |
- Präparate 15 min im Exsikkator unter Vakuum trocknen
|
-
Sonde in der Hybridisierungslösung für - Hybridisierungslösung schnell auf Eis überführen und 5 min abkühlen lassen - Lösung auf Vortex-Schüttler mischen, in Tischzentrifuge abzentrifugieren und bis zum Auftropfen auf Eis stehen lassen |
- pro OT 10 µl Hybridisierungslösung auftropfen, DG möglichst luftblasenfrei auflegen und mit Fixogum abdichten - OT in Wärmeschrank legen und Fixogum 30 min trocknen lassen - OT in feuchte Kammer legen und darin über Nacht bei 37°C hybridisieren |
Post-Hybrisisierungs-Waschung
Die Hybridisierung mit endmarkierten, synthetischen Oligonukleotiden erfolgte in Anlehnung an die Hybridisierung mit klonierten DNA-Sequenzen. Die geringere Länge der Oligonukleotide erfordert gegenüber den längeren klonierten Sequenzen Änderungen bei der Schmelz- und Hybridisierungstemperatur.
Die Schmelztemperaturen (Tm) der verschiedenen 5'-endmarkierten Oligonukleotide (Tabelle 5) wurden nach einer empirisch ermittelten Formel (3) für Sequenzen kürzer als 20 Nukleotide berechnet und die Oligonukleotide bei (Tm-5°C) bis (Tm-10°C) hybridisiert (Suggs et al. 1981; Thein und Wallace 1986).
Formel zur Berechnung von Tm von Oligonukleotiden in 1 M Na+:
Tabelle 5: Schmelz (Tm)- und Hybridisierungstemperaturen (Th) der verwendeten 5'-endmarkierten Oligonukleotid-Sonden. Werte in Klammern kennzeichnen die Temperaturen unter denen die Hybridisierung wiederholt wurden.
Oligonukleotid |
Tm[°C] |
Th[°C] |
|---|---|---|
(C)16 |
64 |
59 (50,40) |
(AT)8 |
32 |
27 |
(AG)8 |
48 |
43 |
(CA)8 |
48 |
43 |
(GC)8 |
64 |
59 (50,40) |
(AAT)5 |
30 |
25 |
(AAG)5 |
40 |
35 |
(CAC)5 |
50 |
45 |
(GCC)5 |
60 |
55 (40) |
(AATG)4 |
40 |
35 |
(GACA)4 |
48 |
43 |
(GATA)4 |
40 |
35 |
M13con |
50 |
45 |
33con |
54 |
49 |
Da die Hybridisierungen mit (C)16 und (GC)8 bei 59°C keine Signale zeigten, wurden die beiden Oligonukleotide in zusätzlichen Versuchen bei 40° und 50°C hybridisiert.
Chemikalien und Lösungen
Oligonukleotid-Sonde: 5'-endmarkiertes Oligonukleotid, 10 pmol/µl
Oligo-Hybridisierungsmix bestehend aus: 5 x SSPE, 5 x Denhardts, 0,1% SDS, 1%
BSA
20 x SSPE: 3 M NaCl, 0,2 M Na2PO4 H2O, 0,02 M
Na2 EDTA, pH 7,4
50 x Denhardts: 5 g Ficoll (Typ 400), 5 g Polyvinylpyrrolidon
6 x SSC: 300 ml 20 x SSC add 1000 ml
20 x SSC: 3 M NaCl, 0,3 M Na3-Citrat 2H20, pH
7,0
Durchführung
Denaturierung und Hybridisierung
Die Hybridisierung des getailten Oligonukleotids für die Arabidopsis-Telomer-Sequenz basiert auf dem Protokoll von Schwarzacher und Heslop-Harrison (1991) und erfolgte wie von Gortner et al. (1998) für Cicer arietinum beschrieben.
Das getailte Mikrosatelliten-Motiv (C)16 hingegen wurde unter den Bedingungen für die 5'-endmarkierte Oligonukleotide hybridisiert und gewaschen, was für ein getailtes Hexadekamer mit 100% GC-Gehalt als niedrigstringent anzusehen ist.
Chemikalien und Lösungen
20 x SSC: 3 M NaCl, 0,3 M Na3-Citrat 2H20, pH
7,0
50% (w/v) Dextransulfat in Aqua dest.
10% (w/v) SDS
getailtes Oligonukleotid, 7-10 pmol/µl
Poly(A)-Lösung, 10 mg/ml (Bohringer Mannheim)
Formamid-Lösung: 25% (v/v) deionisiertes Formamid (Biomol) in 2 x SSC
Durchführung
Denaturierung und Hybridisierung
Die Hybridisierungslösung für 10 µl/OT setzte sich zusammen aus: 25% (v/v) deionisiertes Formamid, 2 x SSC, 10% (w/v) Dextransulfat, 0.5% (v/v) SDS, 1 pmol/µl getailtes Oligonukleotid und 0,1 mg/µl Poly(A)-Lösung.
Alle anderen Arbeitsschritte der Denaturierung und Hybridisierung erfolgten wie unter Abschnitt 2.6.1.1 angegeben.
Post-Hybridisierungs- und Stringenz-Waschung
Um die hybridisierten Sondenmoleküle nachzuweisen, wurden sie entweder direkt oder indirekt mit Hilfe des Avidin/Biotin-Systems oder mit Anti-Digoxigenin- und weiteren Fluorochrom-konjugierten Antikörpern detektiert.
Für die direkte Detektion ist nach der Hybridisierung und Stringenzwaschung kein zusätzlicher Arbeitsschritt notwendig. Die Präparate können umgehend untersucht werden.
Die beiden Moleküle Avidin und Biotin haben eine sehr hohe Bindungsaffinität und eignen sich daher besonders gut als histochemisches Nachweissystem. Biotin-markierte Sonden lassen sich durch Inkubation mit Avidin-Molekülen sichtbar machen, indem letztere mit einem Fluoreszenzfarbstoff konjugiert sind. Die am häufigsten verwendeten Fluorochrome sind FITC (grün/gelb), TRITC (rot) oder Cy3 (rot).
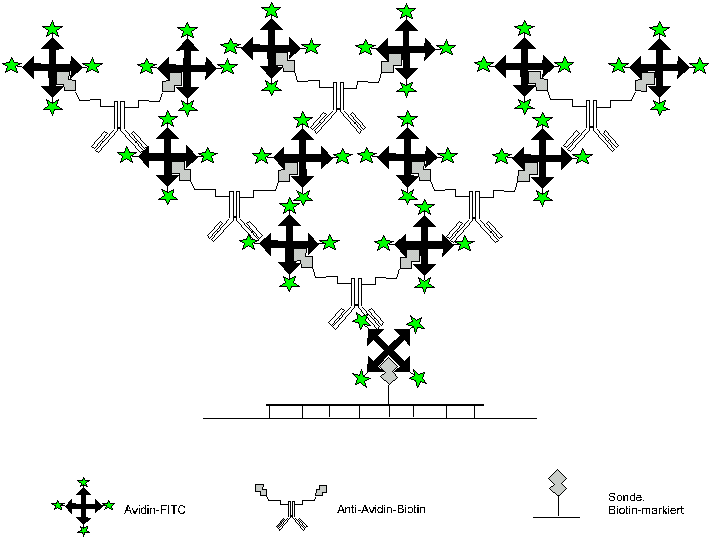
Bei Zielsequenzen, die nur in geringer Kopienzahl vorliegen, ist es notwendig, die Intensität der Fluoreszenz am Ort der Hybridisierung (Signal) zu erhöhen. Dazu inkubiert man nach der ersten Lage Fluorochrom-gekoppeltem Avidin zusätzlich mit Biotin-makierten Anti-Avidin-Antikörpern und dann wiederum mit Fluorochrom-gekoppeltem Avidin, und erhält so eine Verstärkung der Fluoreszenzsignale, was als Signalamplifikation bezeichnet wird (Pinkel et al. 1986).
Zur Detektion der mittelreptitiven Gene der 18S-25S RNA genügte eine einfache Amplifikation, bestehend aus 2 Lagen FITC-konjugiertem Avidin. Für den Nachweis der nur in geringer Kopienzahl vorliegenden Phaseolin-Gene hingegen waren bis zu 4 Lagen FITC-gekoppeltem Avidin notwendig (Abbildung 2).

Abbildung 2: Detektionsschema der Signalamplifikation im Avidin/Biotin-System (4 Lagen)
Material, Chemikalien und Lösungen
BT-Puffer: 0,05% (v/v) Tween 20® (Fluka) in 0,1 M
Natriumhydrogencarbonat pH 8,3
Blockierungs-Lösung-1: 5% (w/v) BSA (Sigma, A-7906) in BT-Puffer
Avidin-FITC-Lösung-1:
Avidin DN-FITC (Vector) 1:100 verdünnt (5
µg/ml) in Blockierungs-Lösung
alternativ wurde auch
ExtrAvidin-FITC (Sigma) oder ExtrAvidin-TRITC (Sigma) verwendet
Blockierungs-Lösung-2: 5% (w/v) Ziegenserum in BT-Puffer
Anti-Avidin-Lösung:
Anti-Avidin D (Vector) 1:50 verdünnt (10
µg/ml) in Blockierungs-Lösung-2
Avidin-FITC-Lösung-2:
Avidin DN-FITC (Vector) 1:50 verdünnt (10
µg/ml) in Blockierungs-Lösung-1
alternativ wurde auch
ExtrAvidin-FITC (Sigma) oder ExtrAvidin-TRITC (Sigma) verwendet
Durchführung
Detektion und Signalamplifikation im Avidin/Biotin-System erfolgte nach Pinkel et al. (1986).
Die Waschschritte, Blockschritte und Antikörper-Inkubationen wurden in einer feuchten Kammer bei RT (Oligonukleotide) bzw. bei 37°C (klonierte Sequenzen) durchgeführt. Die Volumenangaben verstehen sich pro Objektträger (OT).
1. Lage: Detektion mit Avidin-FITC
Anti-Avidin-Biotin
Die Signalamplifikation wurde je nach Signal-Hintergrund-Verhätnis bis zu zweimal wiederholt, bevor die Chromosomen mit DAPI gefärbt wurden (vgl. Abschnitt 2.7).
Zur Detektion von Sonden, die mit Digoxigenin (DIG) markiert sind, verwendet man generell Anti-DIG-Antikörper. Handelt es sich bei der nachzuweisenden Zielsequenz um eine hoch- oder mittel-repetitive Sequenz oder ist die Länge des verwendeten Sondenmoleküls relativ groß (z. B. Cosmid- oder YAC-Klone), reicht eine Lage Fluorochrom-gekoppelter Anti-DIG-Antikörper meist aus.
Liegen die Zielsequenzen dagegen nur in geringer Kopienzahl vor oder werden sehr kurze Sondenmoleküle verwendet, ist eine Vertstärkung des Signals notwendig. Im einfachsten Fall verwendet man dazu Fluorochrom-gekoppelte Anti-DIG-Antikörper, z. B. aus der Maus, und verstärkt das Signal mit einer zweiten Inkubation mit Fluorochrome-konjugiertem Anti-Maus-Antikörper. Durch Einfügen eines Digoxigenin-gekoppelten Antikörpers läßt sich eine dreischichtige Kaskade bestehend aus Maus-Anti-DIG-, Anti-Maus-DIG- und Fluorochrom-gekoppeltem Anti-DIG-Antikörper (Abbildung 3) aufbauen. Diese Kombination von Antiköpern wird als Set unter dem Namen "Fluorescent Enhancer Set for DIG Detection" kommerziell von der Firma Boehringer Mannheim angeboten. Prinzipiell ließe sich die Kaskade durch zwischgeschaltete, abwechselnde Lagen von Maus-Anti-DIG und DIG-Anti-Maus und noch weiter ausbauen. Da die notwendigen Antikörper aber deutlich konzentrierter eingesetzt werden als die entsprechenden Agenzien beim Avidin/Biotin-System und darüber hinaus vergleichsweise teuer sind, wurde darauf verzichtet.
Für den Nachweis der Telomer-Sequenz mit Hilfe des Digoxigenin-getailten Oligonukleotids wurde, wie in der Arbeit von Gortner et al. (1998) beschrieben, eine dreischichtige Detektions-Kaskade verwendet (Abbildung 3).
Abbildung 3: Dreischichtiges Detektionsschema für Digoxigenin-markierte Sonden
Material, Chemikalien und Lösungen
DIG-Puffer-1: 100 mM Maleinsäure, 150 mM NaCl, pH 7,5
DIG-Puffer-1/Tween: 0.2% (v/v) Tween 20® (Fluka) in DIG-Puffer-1
DIG-Puffer-2: 0.5 % (w/v) DIG-Blocking Reagenz (Boehringer Mannheim) in
DIG-Puffer-1
Maus-Anti-Digoxigenin -Lösung:
Maus-Anti-Digoxigenin-AK (monoklonal)
1:300 verdünnt (0,3 µg/ml) in DIG-Puffer-2
Anti-Maus-DIG-Lösung:
Digoxigenin-gekoppelter Anti-Maus-AK
(F(ab')2-Fragment) 1:8 verdünnt (25 µg/ml) in DIG-Puffer-2
Anti-DIG-FITC-Lösung:
FITC-gekoppelter Anti-Digoxigenin-AK
(Fab-Fragment) 1:8 (25 µg/ml) in DIG-Puffer-2
Durchführung
Waschschritte und Antikörper-Inkubationen wurden in einer feuchten Kammer bei RT durchgeführt. Die Volumenangaben verstehen sich pro Objektträger (OT).
Präparate 3 min in DIG-Puffer-1 äquilibrieren
Maus-Anti-DIG
Anschließend werden die Chromosomen, wie im Abschnitt 2.7 beschrieben, mit DAPI gefärbt.
Um die sehr kurzen Sequenzen von Oligonukleotiden nachzuweisen, wurde ein von Dr. T. Becker (persönliche Mitteilung) vorgeschlagenes "Hybrid"-System zur Signalamplifikation von DIG-markierten Sonden (Köhler 1992) eingesetzt. Dazu erfolgte ein erster Inkubationssschritt mit Maus-Anti-DIG-Antikörper und ein zweiter Schritt mit Biotin-gekoppeltem Anti-Maus-Antikörper. Darauf aufbauend fährt man gemäß dem Avidin-Biotin-Amplifikationsschema (Abschnitt 2.6.2.2) fort. Damit verbindet man DIG-markierte Sonden mit der einfachen und kostengünstigen Signalamplifikation des Avidin-Biotin-Systems. Diese Kombination soll im folgenden als "Digoxigenin-Biotin-Amplifikations-System" oder kurz DBA-System bezeichnet werden (Abbildung 4).
Abbildung 4: Schema des Digoxigenin-Biotin-Amplifikations-System (DBA-System)
Material, Chemikalien und Lösungen
Maus-Anti-DIG-Lösung:
Maus-Anti-Digoxigenin-AK (monoklonal) 1:300
verdünnt (0,3 µg/ml) in DIG-Puffer-2
Anti-Maus-Biotin-Lösung:
Biotin-gekoppelter Anti-Maus-AK,
F(ab')2-Fragment, 1:300 verdünnt (1,7 µg/ml) in DIG-Puffer-2
Avidin-FITC-Lösung-1:
Avidin DN-FITC (Vector) 1:100 verdünnt (5
µg/ml) in DIG-Puffer-2
Anti-Avidin-Lösung:
Anti-Avidin D (Vector) 1:50 verdünnt (10
µg/ml) in DIG-Puffer-2
Avidin-FITC-Lösung-2:
Avidin DN-FITC (Vector) 1:50 verdünnt (10
µg/ml) in DIG-Puffer-2
Durchführung
Das Protokoll des DBA-Systems entspricht dem für die dreischichtigen Signalamplifikation für DIG-markierte Sonden (Abschnitt 2.6.2.4), aber mit folgenden Antikörpern:
Signalamplifikation
Die Signalamplifikation wurde je nach Signal-Hintergrund-Verhätnis, einmal wiederholt, und die Chromosomen anschließend mit DAPI gefärbt (vgl. Abschnitt 2.7).




